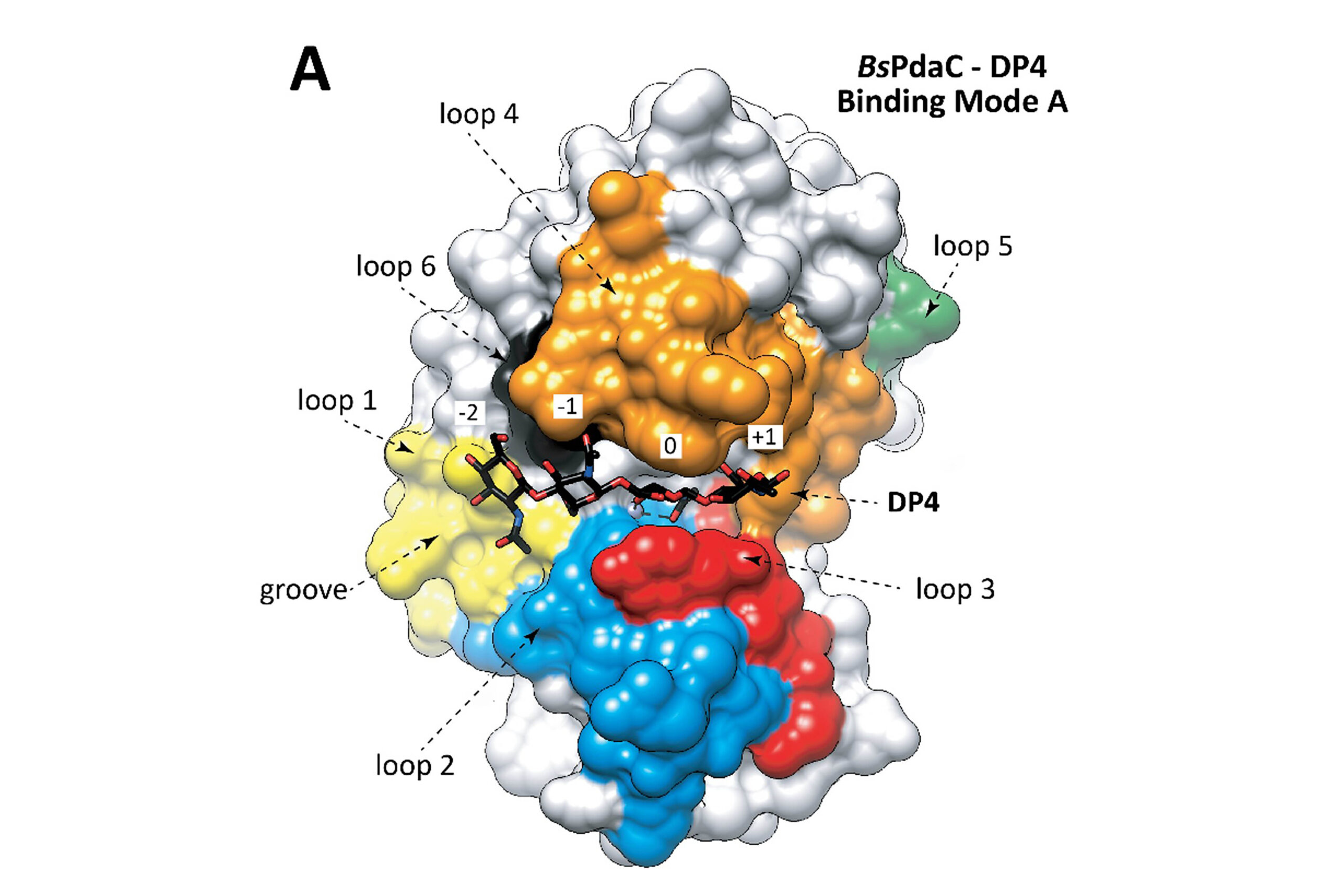
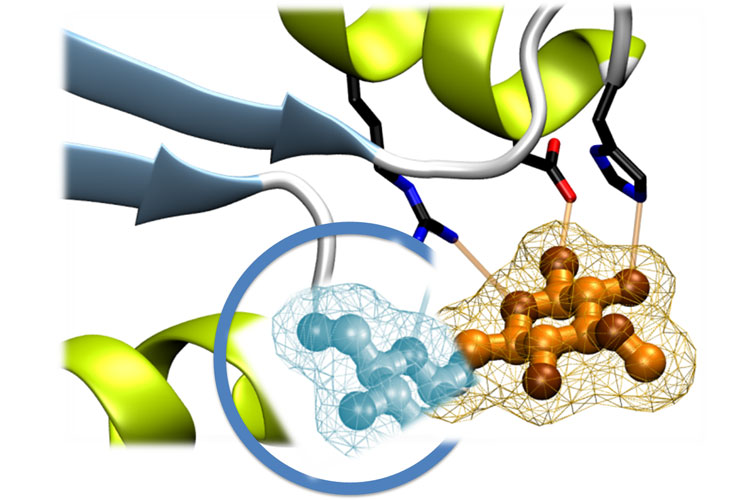
Els enzims catalitzen un gran nombre de reaccions bioquímiques amb notable eficàcia i especificitat, gràcies a la seva capacitat per a reorganitzar dinàmicament l’entorn catalític de les reaccions sobre substrats naturals. Malgrat tot, per a transformacions químiques no convencionals, les interaccions enzim-substrat no estan optimitzades i existeix molt de marge per a la millora de l’eficàcia catalítica. Existeix una creixent demanda d’enzims eficients, selectius i estables per a la seva aplicació en processos de transformació química sostenibles. Aquesta demanda ha impulsat grans avenços a l’enginyeria enzimàtica computacional, que l’han convertit en una revolució científica que permet el desenvolupament de nous biocatalitzadors, com per exemple per a la degradació de plàstics o per a la síntesi d’antibiòtics.
Disseny d’enzims amb aplicacions en biotecnologia i biomedicina

Des del Laboratori de Bioquímica d’IQS (laboratori multidisciplinari del GQBB – Grup de Química Biològica i Biotecnologia que lidera el Dr. Antoni Planas), la unitat de Bioinformàtica i Modelització Molecular, que coordina el Dr. Xevi Biarnés, treballa en el desenvolupament i l’aplicació de mètodes de biologia computacional i simulació, basats en sistemes d’intel·ligència artificial, Data science i càlculs d’altes prestacions. Una de les eines que ha creat aquesta unitat és el protocol BINDSCAN per al disseny i optimització d’enzims. Aquesta eina permet optimitzar la seqüència d’un biocatalitzador de partença, per tal de millorar el seu rendiment i eficiència de reacció sobre substrats no necessàriament naturals.
Dins del grup GQBB, un dels focus de recerca són les glicosil hidrolases (GH), uns enzims que catalitzen la ruptura de l’enllaç glicosídic en glicans i glicoconjugats. La reenginyeria de la funció enzimàtica de les GH ha permès a aquests investigadors produir carbohidrats d’alt valor afegit, com per exemple prebiòtics per a llets de fórmula, polisacàrids per a nanomaterials o estimulants de plantes, entre d’altres.
En aquest ampli context, i amb l’objectiu d’ampliar les capacitats de predicció de l’eina BINDSCAN, el Dr. Aitor Vega Sánchez ha dut a terme la seva tesi doctoral sota el títol BINDSCAN: un protocol computacional per a l’enginyeria de glicosil hidrolases, que ha estat dirigida pel Dr. Xevi Biarnés Fontal i pel Dr. Antoni Planas Sauter, al Departament de Bioenginyeria d’IQS.
Nou protocol computacional basat en l’anàlisi mutacional massiu d’enzims
La tesi del Dr. Vega ha establert, per primera vegada, un protocol computcaional robust que permet quantificar la contribució electrostàtica dels enzims a la seva eficiència catalítica. Des de l’anàlisi mutacional massiva de seqüències proteiques, aquest enfocament identifica posicions clau que poden ser modificades per tal de millorar propietats funcionals, com són el reconeixement de nous lligands o el rendiment catalític, ampliant d’aquesta manera les capacitats de l’eina BINDSCAN per al disseny de biocatalitzadors per a transformacions químiques.
Com a part d’aquesta recerca, s’ha desenvolupatuna versió avançada de l’eina BINDSCAN, que integra mètriques innovadores basades en afinitat d’unió i potencial electroestàtic, juntament amb una interfície gràfica que facilita la interpretació dels resultats.
Aquesta tesi representa un avenç significatiu a la integració entre el disseny computacional i la validació experimental, i amplia el potencial de l’enginyeria enzimàtica en aplicacions industrials i biotecnològiques.
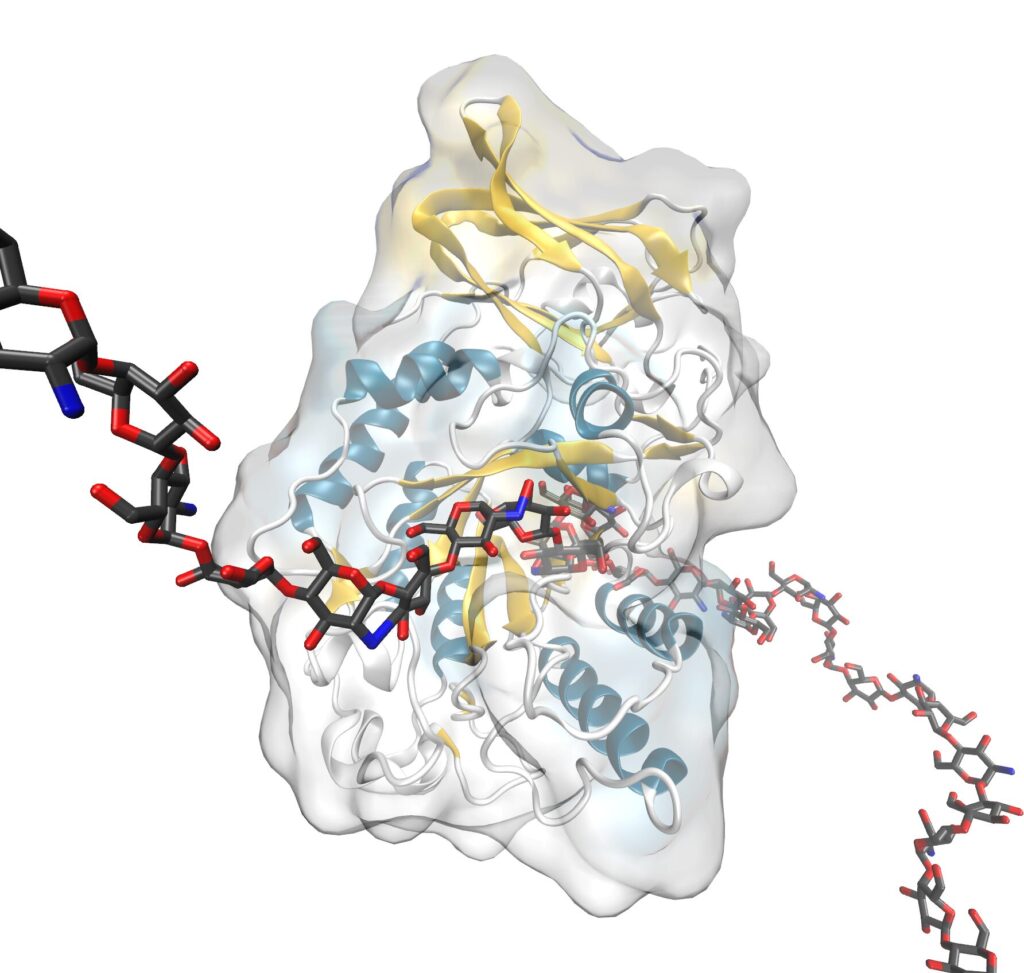
Dins del mateix grup GQBB, aquest nou protocol BINDSCN ha estat aplicat per a l’optimització de la síntesi enzimàtica d’oligosacàrids de llet materna humana (HMOs), a partir del redisseny de l’enzim bacterià lacto-N-biosidasa de Bifidobacterium bifidum, per a la síntesi de lacto-N-tetraosa – LNT, el principal HMO de classe I.
A més, BINDSCN ha estat aplicat també per al disseny d’altres enzims sintètics, en col·laboració amb investigadors de l’Institut de Biotecnologia de Toulouse (TBI), aconseguint la producció d’oligosacàrids d’alt valor afegit d’origen vegetal.
Publicacions relacionades
Aitor Vega, Antoni Planas, Xevi Biarnés, Electrostatic potential as a reactivity scoring function in computer-assisted enzyme engineering, 2025, The FEBS Journal.
Aitor Vega, Antoni Planas, Xevi Biarnés, A practical guide to computational tools for engineering biocatalytic properties, Int. J. Mol. Sci. 2025, 26(3), 980.
Bastien Bissaro, Julien Durand, Xevi Biarnés, Antoni Planas, Pierre Monsan, Michael J. O’Donohue, Régis Fauré, Molecular Design of Non-Leloir Furanose-Transferring Enzymes from an α-l-Arabinofuranosidase: A Rationale for the Engineering of Evolved Transglycosylases. ACS Catalysis 2015, 5(8), 4598–4611
Aquesta recerca s’ha dut a terme en el marc dels projectes GLYCODESIGN (PID2019-104350RB-I00) i GLYCOENGIN (PID2022-138252OB-I00), finançats pel Ministeri de Ciència, Innovació i Universitats – Agència Estatal d’Investigació, dins de la convocatòria dels Programes Estatals de Generació de Coneixement i Enfortiment Científic i Tecnològic del sistema R+D+I orientada als reptes de la societat. També, ha comptat amb finançament addicional de l’Obra Social “la Caixa”, des del programa d’ajudes a la recerca de la Universitat Ramon Llull (Grant: BINDSCAN 2.0’2020).