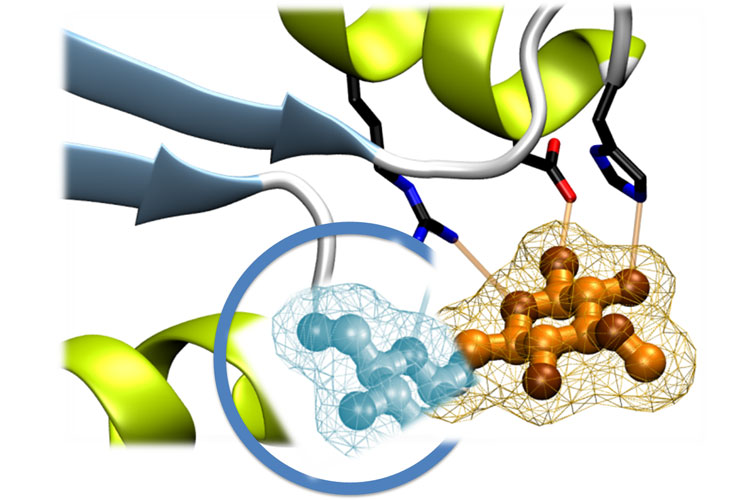
Las enzimas catalizan un gran número de reacciones bioquímicas con notable eficacia y especificidad, gracias a su capacidad para reorganizar dinámicamente el entorno catalítico de las reacciones sobre sustratos naturales. Sin embargo, para transformaciones químicas no convencionales, las interacciones enzimas-sustrato no están optimizadas y existe mucho margen para la mejora de la eficacia catalítica. Existe una creciente demanda de enzimas eficientes, selectivas y estables para su aplicación en procesos de transformación química sostenibles. Esta demanda ha impulsado grandes avances en la ingeniería enzimática computacional, que la han convertido en una revolución científica, permitiendo el desarrollo de nuevos biocatalizadores, como por ejemplo para la degradación de plásticos o la síntesis de antibióticos.
Desde el Laboratorio de Bioquímica de IQS (laboratorio multidisciplinar del GQBB – Grupo de Química Biológica y Biotecnológica que lidera el Dr. Antoni Planas), la unidad de Bioinformática y Modelización Molecular, que coordina el Dr. Xevi Biarnés, trabaja en el desarrollo y la aplicación de métodos de biología computacional y simulación, basados en sistemas de inteligencia artificial, Data sciencey cálculos de altas prestaciones. Una de las herramientas que ha creado esta unidad es el protocolo BINDSCAN para el diseño y optimización de enzimas. Esta herramienta permite optimizar la secuencia de un biocatalizador de partida, a fin de mejorar su rendimiento y eficiencia de reacción sobre sustratos no necesariamente naturales.
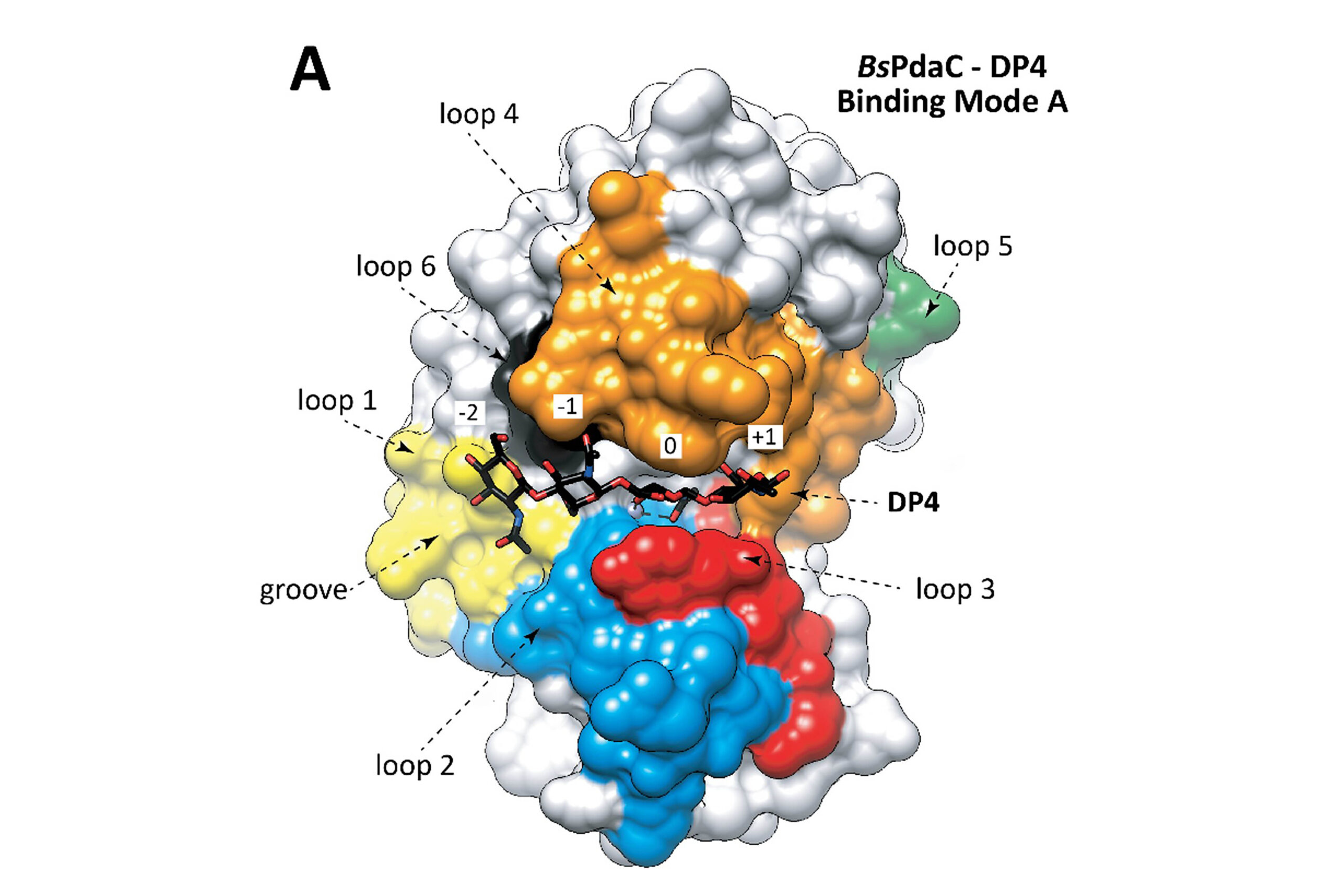
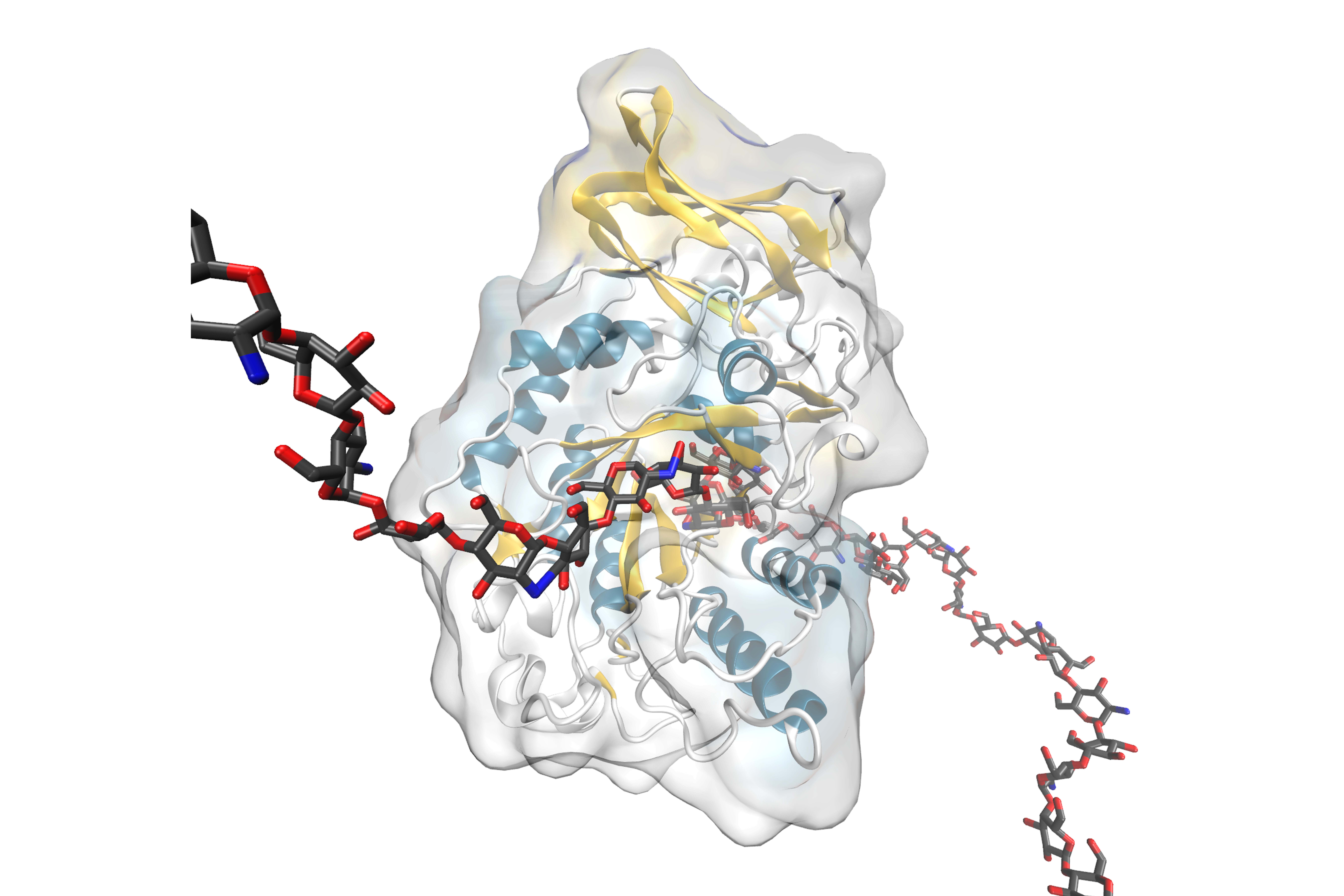
Dentro del grupo GQBB, uno de sus focos de investigación son las glicosil hidrolasas (GH), unas enzimas que catalizan la ruptura del enlace glicosídico en glicanos y gliconjugados. La reingeniería de la función enzimática de las GH ha permitido a estos investigadores producir carbohidratos de alto valor añadido, como por ejemplo prebióticos para leches de fórmula, polisacáridos para nanomateriales, o estimulantes de plantas, entre otros.
En este amplio contexto, y con el objetivo de ampliar las capacidades de predicción de la herramienta BINDSCAN, el Dr. Aitor Vega Sánchez ha llevado a cabo su tesis doctoral bajo el título BINDSCAN: un protocolo computacional para la ingeniería de glicosil hidrolasas, que ha sido dirigida por el Dr. Xevi Biarnés Fontal y por el Dr. Antoni Planas Sauter en el Departamento de Bioingeniería de IQS.
Nuevo protocolo computacional basado en el análisis mutacional masivo de enzimas
La tesis del Dr. Vega ha establecido, por primera vez, un protocolo computacional robusto que permite cuantificar la contribución electrostática de los enzimas a su eficiencia catalítica. A través del análisis mutacional masivo de secuencias proteicas, este enfoque identifica posiciones clave que pueden ser modificadas para mejorar propiedades funcionales, como el reconocimiento de nuevos ligandos o el rendimiento catalítico, ampliando así las capacidades de la herramienta BINDSCAN para el diseño de biocatalizadores para transformaciones químicas.
Como parte de esta investigación, se ha desarrollado una versión avanzada de la herramienta BINDSCAN, que integra métricas innovadoras basadas en afinidad de unión y potencial electrostático, junto con una interfaz gráfica que facilita la interpretación de los resultados.
Esta tesis representa un avance significativo en la integración entre el diseño computacional y la validación experimental, y amplía el potencial de la ingeniería enzimática en aplicaciones industriales y biotecnológicas.
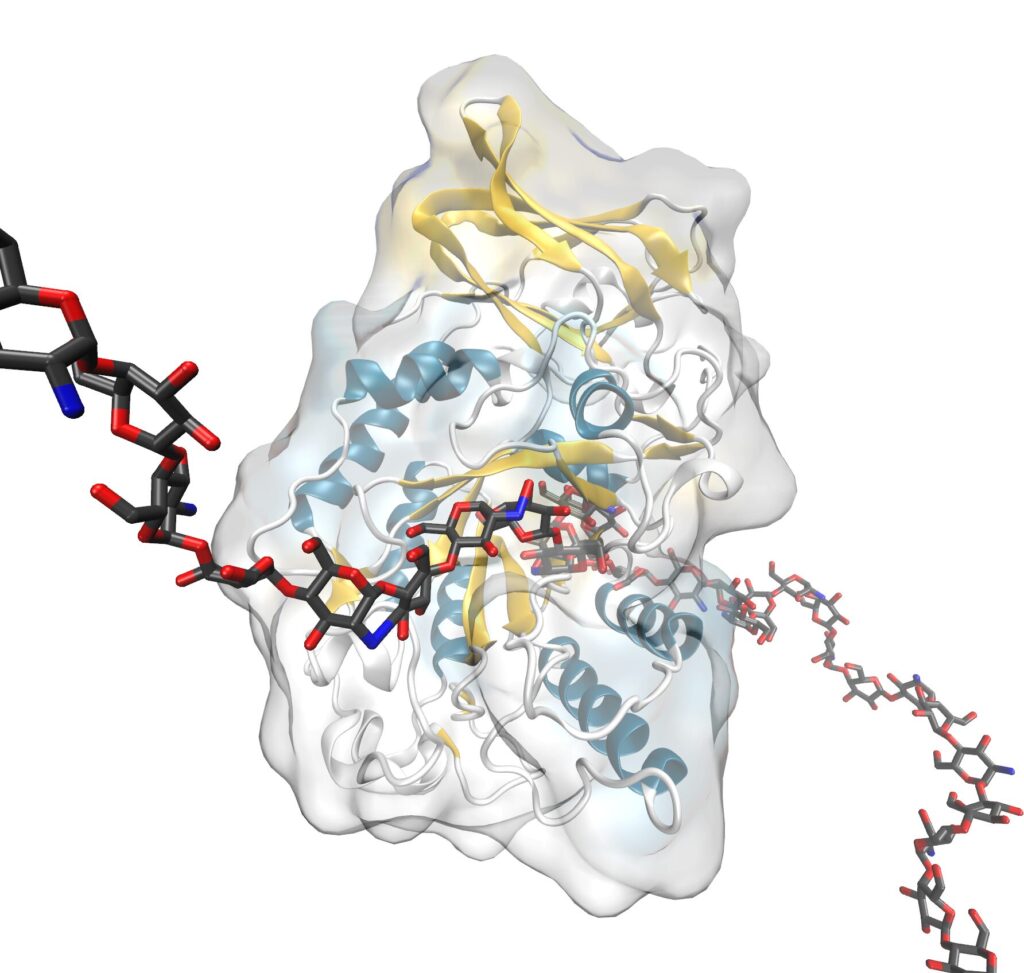
Dentro del propio grupo GQBB, este nuevo protocolo BINDSCAN ha sido aplicado para la optimización de la síntesis enzimática de oligosacáridos de leche materna humana (HMOs), a partir del rediseño de la enzima bacteriana lacto-N-biosidasa de Bifidobacterium bifidum, para la síntesis de lacto-N-tetraosa – LNT, el principal HMO de clase I.
Además, BINDSCAN ha sido aplicado también para el rediseño de otras enzimas sintéticas, en colaboración con investigadores del Instituto de Biotecnología de Toulouse (TBI), permitiendo la producción de oligosacáridos de alto valor añadido de origen vegetal.
Publicaciones relacionadas
Aitor Vega, Antoni Planas, Xevi Biarnés, Electrostatic potential as a reactivity scoring function in computer-assisted enzyme engineering, 2025, The FEBS Journal.
Aitor Vega, Antoni Planas, Xevi Biarnés, A practical guide to computational tools for engineering biocatalytic properties, Int. J. Mol. Sci. 2025, 26(3), 980.
Bastien Bissaro, Julien Durand, Xevi Biarnés, Antoni Planas, Pierre Monsan, Michael J. O’Donohue, Régis Fauré, Molecular Design of Non-Leloir Furanose-Transferring Enzymes from an α-l-Arabinofuranosidase: A Rationale for the Engineering of Evolved Transglycosylases. ACS Catalysis 2015, 5(8), 4598–4611
Esta investigación se ha llevado a cabo en el marco de los proyectos GLYCODESIGN (PID2019-104350RB-I00) y GLYCOENGIN (PID2022-138252OB-I00), financiados por el Ministerio de Ciencia, Innovación y Universidades – Agencia Estatal de Investigación, dentro de la convocatoria de los Programas Estatales de Generación de Conocimiento y Fortalecimiento Científico y Tecnológico del sistema de I+D+i orientada a los retos de la sociedad. También ha contado con financiación adicional de la Obra Social “laCaixa”, desde el programa de ayudas a la investigación de la Universidad Ramon Llull (Grant: BINDSCAN 2.0’2020).